Есть такая область биологии — синтетическая биология. Вообще, ей уже лет десять, она развивается очень бурно, время от времени какие-то новости прорываются в научно-популярные издания, но что-то это всё мимо меня проскакивало. А тут вдруг наткнулся, почитал несколько статей — и очень впечатлился.Главная идея синтетической биологии — синтезировать на генетическом уровне вещи, которые то ли не появились, то ли не закрепились в эволюции жизни на Земле. Под словом "вещи" может иметься в виду как функция, так и что-то материальное — например, новые белки или даже новые аминокислоты, из которых можно строить совершенно новые типы белков. И из этих новых "кирпичиков" биологи-синтетики пытаются построить, нет, даже так — запрограммировать — новые варианты жизни. Это как бы генетический инженеринг, но на совершенно новом уровне — здесь не пересаживают ген одного организма другому, здесь пытаются с нуля "рассчитать" новый способ жизнедеятельности и внедрить его в реально живущую клетку.Какие функции тут можно реализовать и как? Пока самой распространенной "игрой" является программирование новых, не существовавших в природе молекулярно-генетических "часов" в клетках (чаще всего, это бактерии E.coli). Вот классический пример (Nature, 2000): в клетку запускают три белка (A, B, C), которые могут вырабатываться самой клеткой, но которые подавляют экспрессию друг друга по цепочке: A подавляет B, B подавляет C, C подавляет A. В результате возникает петля обратной связи — но с задержкой по времени. И этого уже достаточно, чтобы в размножающейся колонии бактерий начались колебания концентрации этих молекул, что можно отслеживать напрямую по зеленому флуоресцентному белку (побочному продукту на одной из стадий цикла). Получается такая картинка: 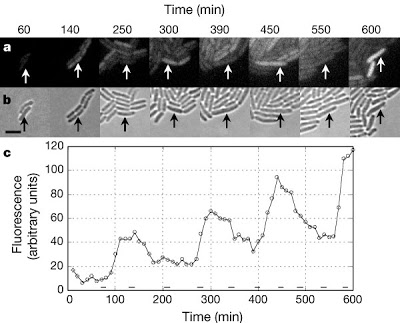 Обратите внимание — период колебаний здесь составляет часы, что в несколько раз больше периода деления клеток. Получается, информация о том, в какой фазе колебания мы находимся, генетически передается из поколения в поколение.Поначалу у таких работ были недостатки — далеко не все клетки вовлекались в колебание, наблюдался сильный разброс откликов по всей популяции, да и с течением времени разные клетки сбивались с ритма или начинали подзабывать фазу. Однако с этими проблемами постепенно справились. В 2008 году в работе A fast, robust and tunable synthetic gene oscillator отклик был сильный, устойчивый и однородный, а буквально месяц назад была опубликована работа A synchronized quorum of genetic clocks, в которой клетки, общаясь друг с другом, успешно синхронизировали по всей популяции свои новоприобретенные генетические часы.Отдельно подчеркну роль теорфизики. За 6 лет до работы 2008 года в Phys.Rev.Lett. была публикована работа Synthetic Gene Network for Entraining and Amplifying Cellular Oscillations, в которой строилась модель подобных осцилляций и изучалась их фазовая диаграмма (например, при изменении силы петель обратной связи). В работе 2008 года опыт этого моделирования был принят к сведению (один из авторов, кстати, участвовал в обоих работах).Это, конечно, только одна из возможностей. Сейчас из набора таких транскприпционных факторов уже умеют создавать элементы логических схем и вроде бы недавно даже внедрили в ту же E.coli настоящий цифровой регистр, который "считал" количество событий деления. В общем, тут открываются головокружительные перспективы — см. например (довольно старую) популярную статью Синтетическая жизнь. Правда, это всё делать не так просто — о технических трудностях этих работ см. недавний материал из Nature: Пять горьких истин синтетической биологии. Это конечно впечатляет, но это еще далеко не всё. Дальше — круче.Предположим, нам хочется создать новые белки, построенные не только на стандартных 22-х, но и на каких-то новых аминокислотах. В принципе, другие аминокислоты есть, только в природе не предусмотрена возможность их кодирования в РНК. Как сделать так, чтоб рибосома их всё же использовала при синтезе белка?Один из вариантов — заставить рибосому мутировать так, чтоб она на каком-то не сильно важном триплете "ошибалась" — вставляла другую аминокислоту. В принципе, такие работы были, но как-то вяло всё шло. А неделю назад была опубликована статья Encoding multiple unnatural amino acids via evolution of a quadruplet-decoding ribosome, в которой реализовано совершенно радикальное решение этой проблемы. Авторы этой работы целенаправлено добились такой мутации рибосом, чтобы они считывали генетический код не триплетами, а квадруплетами — т.е. по четыре "буквы" РНК сразу. При этом открывается огромный простор для кодирования сразу кучи новых аминокислот (квадруплет может закодировать 256 комбинаций вместо 64 у триплета).Для примера авторы смогли встроить в белок кальмодулин парочку новых аминокислот, которые затем в пространстве дополнительно соединились друг с другом (образовали циклический кросс-линк), что значительно укрепило трехмерную пространственную структуру белка (см. картинку):
Обратите внимание — период колебаний здесь составляет часы, что в несколько раз больше периода деления клеток. Получается, информация о том, в какой фазе колебания мы находимся, генетически передается из поколения в поколение.Поначалу у таких работ были недостатки — далеко не все клетки вовлекались в колебание, наблюдался сильный разброс откликов по всей популяции, да и с течением времени разные клетки сбивались с ритма или начинали подзабывать фазу. Однако с этими проблемами постепенно справились. В 2008 году в работе A fast, robust and tunable synthetic gene oscillator отклик был сильный, устойчивый и однородный, а буквально месяц назад была опубликована работа A synchronized quorum of genetic clocks, в которой клетки, общаясь друг с другом, успешно синхронизировали по всей популяции свои новоприобретенные генетические часы.Отдельно подчеркну роль теорфизики. За 6 лет до работы 2008 года в Phys.Rev.Lett. была публикована работа Synthetic Gene Network for Entraining and Amplifying Cellular Oscillations, в которой строилась модель подобных осцилляций и изучалась их фазовая диаграмма (например, при изменении силы петель обратной связи). В работе 2008 года опыт этого моделирования был принят к сведению (один из авторов, кстати, участвовал в обоих работах).Это, конечно, только одна из возможностей. Сейчас из набора таких транскприпционных факторов уже умеют создавать элементы логических схем и вроде бы недавно даже внедрили в ту же E.coli настоящий цифровой регистр, который "считал" количество событий деления. В общем, тут открываются головокружительные перспективы — см. например (довольно старую) популярную статью Синтетическая жизнь. Правда, это всё делать не так просто — о технических трудностях этих работ см. недавний материал из Nature: Пять горьких истин синтетической биологии. Это конечно впечатляет, но это еще далеко не всё. Дальше — круче.Предположим, нам хочется создать новые белки, построенные не только на стандартных 22-х, но и на каких-то новых аминокислотах. В принципе, другие аминокислоты есть, только в природе не предусмотрена возможность их кодирования в РНК. Как сделать так, чтоб рибосома их всё же использовала при синтезе белка?Один из вариантов — заставить рибосому мутировать так, чтоб она на каком-то не сильно важном триплете "ошибалась" — вставляла другую аминокислоту. В принципе, такие работы были, но как-то вяло всё шло. А неделю назад была опубликована статья Encoding multiple unnatural amino acids via evolution of a quadruplet-decoding ribosome, в которой реализовано совершенно радикальное решение этой проблемы. Авторы этой работы целенаправлено добились такой мутации рибосом, чтобы они считывали генетический код не триплетами, а квадруплетами — т.е. по четыре "буквы" РНК сразу. При этом открывается огромный простор для кодирования сразу кучи новых аминокислот (квадруплет может закодировать 256 комбинаций вместо 64 у триплета).Для примера авторы смогли встроить в белок кальмодулин парочку новых аминокислот, которые затем в пространстве дополнительно соединились друг с другом (образовали циклический кросс-линк), что значительно укрепило трехмерную пространственную структуру белка (см. картинку):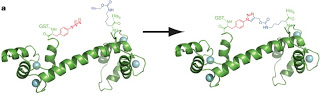 А если вспомнить, что много заболеваний связано именно с неправильным фолдингом белков, то можно легко понять, насколько потенциально перспективными могут стать подобные укрепления.Занятно еще и то, что в принципе обе формы считывания можно организовать параллельно, кодируя гены обоего типа на одной и той же ДНК. Две рибосомы — одна нормальная, другая с квадруплетным считыванием, — будут считывать "свои" гены и не будут понимать "чужие".На закуску несколько ссылок (буду благодарен за хорошие добавления, мембрану и т.п. — не предлагать!):Synthetic systems biology — подборка ключевых статей по этой теме, опубликованных в Nature за последние 10 лет.Журнал Systems and Synthetic Biology, в котором много статей в открытом доступе.Synthetic biology — много обзорных статей в открытом доступе.Synthetic biology projectSyntheticBiology.org — в основном, техническая информация.
А если вспомнить, что много заболеваний связано именно с неправильным фолдингом белков, то можно легко понять, насколько потенциально перспективными могут стать подобные укрепления.Занятно еще и то, что в принципе обе формы считывания можно организовать параллельно, кодируя гены обоего типа на одной и той же ДНК. Две рибосомы — одна нормальная, другая с квадруплетным считыванием, — будут считывать "свои" гены и не будут понимать "чужие".На закуску несколько ссылок (буду благодарен за хорошие добавления, мембрану и т.п. — не предлагать!):Synthetic systems biology — подборка ключевых статей по этой теме, опубликованных в Nature за последние 10 лет.Журнал Systems and Synthetic Biology, в котором много статей в открытом доступе.Synthetic biology — много обзорных статей в открытом доступе.Synthetic biology projectSyntheticBiology.org — в основном, техническая информация.
Нет Ответов